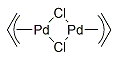π-allyl Pd錯体のモデリング
Winmostarには、有機金属錯体をモデリングするための置換基も収録されています。そこで、有機金属錯体の一例として、[PdCl(η3-allyl)]2(通称π-アリルパラジウム・クロロブリッジダイマー:Fig.1)のモデリングを行ってみることにします。
初期画面より、置換基リストから[-PDH3]を選択して置換し、PdH4を作ります。ここから同じく[-PDH3]を使って水素を置換していき、Fig.2のように井桁状にします。対角線上にある2つのパラジウムから水素を削除し、塩素に置換します(Fig.3)。残ったパラジウムから一個ずつ水素を削除し、[編集(E)]の[原子移動(M)]を使って水素原子を移動、Fig.4のような対称な形にします。これで中心の4員環部分が出来上がりです。
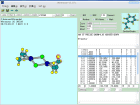
置換基リストの中から、[-PIC3H5]を選択します。これは、π型のアリル配位子という意味です。水素を選択し、[Add]ボタンで置換すると、アリル基が4員環平面と直交した形になります(Fig.5)。これを直すには、少し手間がかかります。まず、画面右上に[All Atoms]というチェックボックスがありますので、それにチェックを入れます。すると、グレーの小さな原子が現れます。これはダミー原子と呼ばれるもので、モデルを組みやすくするために導入するものです。このダミー原子とアリル基の真ん中の炭素を選択し、結合付加ボタンで結合させます。次に、ダミー原子と隣のパラジウム原子を選択し、[編集(E)]の[部分回転(R)]を使って回転させ、ちょうど良い角度にします。最後に、ダミー原子を選択すると、右側の結合パラメータリスト(z-matrix)でもダミー原子(Xx)が選択されますので、結合長を0.8Åに書き換えてリターンキーを押します。これにより、アリル基がパラジウムに近づきます(Fig.6)。あとはダミー原子を削除すればOKです。
反対側も同じように行うことにより、Fig.7のように錯体をモデリングすることができます。